Peroxisomaler Proteinimport
Peroxisomen sind Einzelmembranzellorganellen, die mindestens 50 verschiedene Enzyme enthalten, die an essentiellen Stoffwechselwegen beteiligt sind, einschließlich des oxidativen Abbaus langkettiger Fettsäuren, der Peroxidentgiftung und der Synthese von Etherphospholipiden. Sie sind in Leber, Niere und Zentralnervensystem reichlich vorhanden und können ihre Anzahl und Größe in einzelnen Zell- oder Gewebetypen entsprechend den Umwelteinflüssen dramatisch annehmen. Peroxisomale Membrantransporter müssen aktiv durch Metaboliten wie Elektronenträger (z. B. NAD/H+) und Nukleotide (ATP) oder sogar vollständig gefaltete Proteine durch die peroxisomale Membran navigieren. Es ist bekannt, dass mindestens 33 Pex-Proteine, sogenannte Peroxine, an der Bildung und Aufrechterhaltung von Peroxisomen in Hefen beteiligt sind. Mutationen in einem oder mehreren der zugrunde liegenden PEX-Gene verursachen ein breites Spektrum menschlicher Krankheiten, die als Peroxisomen-Biogenese-Störungen (PBDs) bezeichnet werden. Die Mehrzahl der peroxisomalen Matrixproteine enthält eine C-terminale peroxisomale Zielsequenz Typ 1 (PTS1), die vom löslichen Rezeptorprotein Pex5 erkannt wird. Der Cargo-Pex5-Komplex wird an die peroxisomale Membran abgegeben und soll in Kombination mit dem peroxisomalen Membranprotein Pex14 einen temporären proteinleitenden Kanal bilden. Der PTS1-Import hängt von der Monoubiquitinierung und Gewinnung von Pex5 aus der peroxisomalen Membran ab, vermutlich um den Importzyklus aufrechtzuerhalten. Dieser Recyclingschritt ist ATP-abhängig und wird von den beiden Typ II AAA+ (ATPasen, die mit verschiedenen zellulären Aktivitäten assoziiert sind) Peroxinen Pex1 und Pex6 durchgeführt. ATPasen vom Typ II enthalten zwei ATPase-Domänen hintereinander und ihr aktiver Zustand ist normalerweise ein Hexamer. Pex1 und Pex6 bilden einen heterohexameren Komplex mit 700 kDa, der vorgeschlagen wird, sein Substrat durch Einfädeln durch die zentrale Pore umzugestalten.
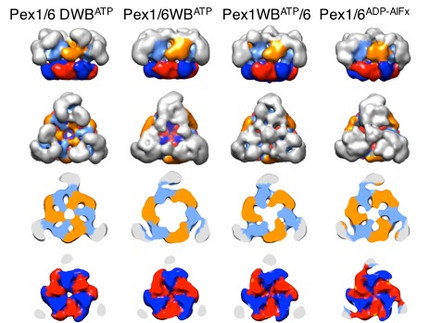
Wir haben niedrig aufgelöste Strukturen von Hefe-Pex1/6-Komplexen in 7 verschiedenen Zuständen des ATPase-Zyklus gelöst und eine umfassende Strukturanalyse seiner nukleotidabhängigen dynamischen Bewegungen angeboten. Die Komplexe sind Trimere von Pex1/6-Dimeren mit einer ausgeprägten Dreiecksform. Die ATPase-Aktivität des Komplexes wird ausschließlich der zweiten AAA + -Domäne (D2) von Pex6 zugeschrieben, während die D2-Domänen von Pex1 und Pex6 Substrat-Interaktionsschleifen enthalten, die für die Komplexfunktion in vivo wesentlich sind. Unsere Karten zeigen, dass eine verlängerte ATP-Bindung an die Pex1/6-D2-Domänen zu einer planaren hexameren Anordnung des D2-Rings führt. Die Nukleotidhydrolyse bewirkt jedoch eine Abwärtsrotation der D2-Domänen. Wir schlagen vor, dass eine ATP-Hydrolyse nur dann auftreten kann, wenn interagierende Motive wie der Argininfinger der benachbarten Domäne links korrekt positioniert sind, um die Kommunikation zu fördern.
Veröffentlichungen:
- Ciniawsky, S., I. Grimm, D. Saffian, W. Girzalsky, R. Erdmann, P. Wendler (2015). Molecular snapshots of the Pex1/6 AAA+ complex in action. Nat Commun. 6:7331. doi:10.1038/ncomms8331 https://www.nature.com/articles/ncomms8331
- Saffert, P., C. Enenkel, P. Wendler (2017). Structure and function of p97 and Pex1/6 Type II AAA+ complexes. Front. Mol. Biosci. 4:33. doi: 10.3389/fmolb.2017.00033 http://journal.frontiersin.org/article/10.3389/fmolb.2017.00033/full
- Schieferdecker A., P. Wendler (2019). Structural mapping of missense mutations in the Pex1/Pex6 complex. International Journal of Molecular Sciences. 20(15), 3756; https://doi.org/10.3390/ijms20153756